2022年2月28日,Genome Biology在線(xiàn)發(fā)表了中國科學(xué)院上海營(yíng)養與健康研究所邵振研究組題為“HyperChIP: identification of hypervariable signals across ChIP-seq or ATAC-seq samples”的方法學(xué)論文,報道了其開(kāi)發(fā)的HyperChIP計算模型。它能對多個(gè)ChIP/ATAC-seq樣本間信號強度差異進(jìn)行統計建模,識別高變信號基因組區域(hypervariable regions,HVRs);一系列下游分析表明,這些表觀(guān)調控信號樣本間差異顯著(zhù)高于背景模型的位點(diǎn)可有效用于揭示被比較樣本集的宏觀(guān)異質(zhì)性結構(疾病亞型或進(jìn)程、組織分化階段、種群遺傳背景等)及其上游調控機制。
真實(shí)人群尤其是癌癥等慢性疾病病人在基因組、轉錄組等很多層面表現出較高的異質(zhì)性。它們緊密聯(lián)系著(zhù)疾病臨床表現等個(gè)體表型差異。但由于表觀(guān)組ChIP-seq和ATAC-seq數據跨樣本定量比較還存在許多公認難題,尚未有能通過(guò)多樣本統計比較來(lái)識別高異質(zhì)性表觀(guān)調控位點(diǎn)的完整計算模型被發(fā)表,造成很多相關(guān)研究只能采用一些經(jīng)驗方法。但是,這些經(jīng)驗分析方法還存在易受技術(shù)因素影響和缺乏統計顯著(zhù)性判據等缺陷,成為制約疾病和正常人群表觀(guān)組異質(zhì)性精準解讀的一個(gè)關(guān)鍵技術(shù)瓶頸。
針對這一點(diǎn),HyperChIP提出使用擬合的ChIP/ATAC-seq信號強度均值-方差關(guān)系來(lái)對每個(gè)位點(diǎn)的觀(guān)測信號方差進(jìn)行標度,并以基因表達數據為參照,指出相比于各種經(jīng)驗統計指標,上述方法獲得的標度方差(scaled variance)能更好地衡量每個(gè)位點(diǎn)表觀(guān)調控信號的樣本間異質(zhì)性水平(圖A) ;然后,在評估其統計顯著(zhù)性時(shí),引入區域篩選(subset selection)和縮尾處理(winsorization)等手段來(lái)控制潛藏真陽(yáng)性位點(diǎn)對參數估計的影響,顯著(zhù)改善了HVR識別的統計功效,實(shí)現對被比較樣本間表觀(guān)組異質(zhì)性的完整統計刻畫(huà)(圖B)。
將HyperChIP應用于42例肺腺癌病人腫瘤的H3K27ac ChIP-seq數據,發(fā)現在識別的HVRs中,很多位點(diǎn)的H3K27ac水平與腫瘤的臨床診斷階段有較強的關(guān)聯(lián)。另一方面,應用它分析由數百例TCGA腫瘤樣本生成的大型pan-cancer ATAC-seq數據集,發(fā)現基于所得HVRs能較好地將不同癌癥類(lèi)型的樣本分開(kāi)。其中一些組織來(lái)源或細胞形態(tài)相近而聚在一起的癌癥樣本,被歸類(lèi)定義為四個(gè)癌癥超類(lèi)型(super class),即腦部腫瘤、消化道腺癌、腎癌和鱗狀細胞癌。進(jìn)一步,通過(guò)構建轉錄因子活性(TF activity)打分模型,發(fā)現了大量被特定癌癥超家族中樣本所共有的轉錄調控因子(圖C)。它們中很多被基因表達數據和其它研究結果支持。例如,TP63被發(fā)現在鱗細胞癌樣本間具有普遍較高的轉錄因子活性,而很多研究已經(jīng)指出它是一個(gè)泛鱗癌致癌因子。
此外,將HyperChIP應用于正常組織ChIP/ATAC-seq數據也能獲得有價(jià)值的發(fā)現。例如,應用它比較著(zhù)床前不同時(shí)間點(diǎn)的小鼠胚胎ATAC-seq數據,對所得的HVRs進(jìn)行主成分分析,發(fā)現第一主成分指征了胚胎的不同發(fā)育階段;進(jìn)一步結合轉錄因子活性分析,可有效發(fā)掘出不同階段所特異的轉錄調控因子。另一方面,應用它比較十余個(gè)不同人類(lèi)個(gè)體的淋巴母細胞系CTCF ChIP-seq數據,對所得的HVRs進(jìn)行主成分分析,發(fā)現能很好地將樣本按個(gè)體所屬種群分開(kāi)(圖D)。
中國科學(xué)院上海營(yíng)養與健康研究所博士生陳浩杰和博士后涂世奇為該論文共同第一作者,邵振研究員和涂世奇為共同通訊作者。復旦大學(xué)生命科學(xué)學(xué)院張一婧研究員、復旦大學(xué)附屬腫瘤醫院孫藝華主任等對該工作的提出和完善也做出了重要貢獻。該工作獲得了國家自然科學(xué)基金委,科技部,中國科學(xué)院的資助。上述研究所使用的所有數據均來(lái)自已公開(kāi)發(fā)表數據。
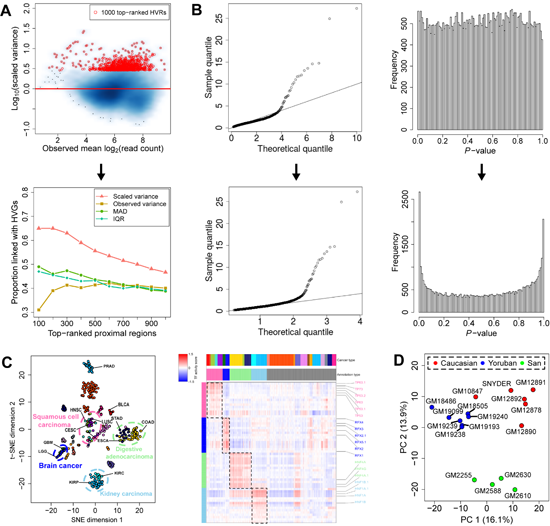
圖:(A) HyperChIP模型以校正了均值-方差關(guān)系的標度方差(scaled variance)為統計指標, 使不同信號強度水平的基因組區域之間的信號差異水平更加可比。(B)通過(guò)引入區域篩選和縮尾處理等手段來(lái)控制潛藏真陽(yáng)性對參數估計的影響,顯著(zhù)改善了HVR識別的統計功效。(C)將HyperChIP應用于TCGA pan-cancer ATAC-seq數據集,基于所得HVRs將部分組織來(lái)源或細胞形態(tài)相近的樣本歸類(lèi)定義為四個(gè)癌癥超類(lèi)型(super class),即腦部腫瘤(Brain cancer)、腎癌(Kidney carcinoma)、消化道腺癌(Digestive adenocarcinoma)和鱗狀細胞癌(Squamous cell carcinoma);下游的轉錄因子活性打分分析,發(fā)現了大量被每個(gè)超家族中腫瘤樣本所共有的轉錄調控因子。(D)比較十余個(gè)不同人類(lèi)個(gè)體的淋巴母細胞系CTCF ChIP-seq數據,發(fā)現基于所得HVRs能很好地區分不同種群個(gè)體的樣本。
論文鏈接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-022-02627-9